Science/Research 詳細
MIT、新しい顕微鏡技術で細胞の3D画像を捉える
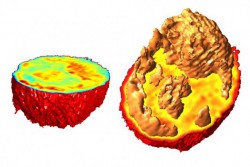
April, 2, 2014, Cambridge--MITの研究グループは、2007年、蛍光マーカーや他のラベルなしで生きた細胞の内部を3Dで詳細に見ることができる顕微鏡を開発した。この技術は、細胞密度と言った重要な特徴も明らかにした。
今回、細胞が微小流体チャネルを流れる際に細胞を撮像するためにその方法を適用した。これは細胞分類システムへの重要な一歩となる。このようなシステムができると、研究者は様々な発展段階で幹細胞を分離したり、あるいは健全な細胞とがん細胞との区別もできるようになる。
他の分類法では、関心のある細胞を目立たせるために蛍光分子を加える必要があるが、そのようなタグは細胞に損傷を与え、細胞が治療利用に不適切となる。
「多くの幹細胞アプリケーションは、分化の異なる段階で細胞の分類が必要になる。これは蛍光染色でできるが、一旦細胞を染色すると使えなくなる。MITのアプローチでは、細胞集団の3D分布についての膨大な量の情報が分類に使える」とMITのレーザ生物医学研究センターの前ポスドク研究者、論文の筆頭著者、Yongjin Sung氏は説明している。
蛍光タグを使う代わりにMITの方法は、細胞の屈折率を分析する。つまり、物質を透過するときに光の速度がどの程度減少するかを計測するのである。全ての物質は特有の屈折率をもっており、この特徴を細胞の体積とともに使って、その質量と密度を計算する。
細胞の個々の部分は、細胞小器官を含め、異なる屈折率を持っているので、このアプローチで得られる情報は、これら細胞の内的構造、核、核小体、核内の構造などの特定にも使える。
新しいMITシステムの重要な特徴は、多くの異なる角度から細胞を照射できる集束レーザビームを用いることである。細胞がビームを横切って流れる際、細胞からの散乱光を分析する。軸外デジタルホログラフィとして知られる技術を用いることで、細胞の各所からの散乱光の振幅と位相を直ちに記録することができる。
現在のシステムは、1秒あたり約10細胞を撮像できるが、研究チームは1秒に数千細胞まで速度を上げたいと考えている。こうなると、幹細胞の分類などのアプリケーションに便利になる。チームは、このシステムを使ってがん細胞の成長、異なる薬剤治療への反応がどうであるかについても理解を深めたいと考えている。
この研究に資金を提供しているのは、国立衛生研究所(NIH)と浜松フォトニクスである。




















