Science/Research 詳細
イリノイ大学、コンピュータ顕微鏡でサブアトミック解像度達成
April, 13, 2018, Champaign--イリノイ大学の研究チームは、分子相互作用を促進する:原子間力およびサブアトミック力をシミュレートできる「コンピュータ顕微鏡」を作製した。研究者によると、この装置は、生命の化学を理解し、大きな分子系をモデル化し、新しい医薬品や産業用物質を開発する研究を簡素化する。
研究成果は、Nature Methodsに発表されている。
研究チームは、分子相互作用のシミュレートに使用される2つのアプローチを統合した。第1は、NAMD(NAnoscale Molecular Dynamics program)として知られるナノスケール分子-ダイナミクスプログラムで、これは古典的な機械学的方法を使って構造をモデル化し、数百万の個々の原子の挙動をシミュレートする。第2プログラムは、サブアトミックの領域をクローズアップし、陽子、中性子、電子の相互作用をシミュレートする。この量子力学的スケールでのモデリングは膨大なコンピューティングパワーを必要とするので、研究チームは大きな分子を古典的、また量子力学的な領域に分ける方法を実行した。これにより、化学結合の生成と破壊など、重要な相互作用に関わる小領域にコンピュータリソースを集中できる。
コンピュータ顕微鏡は、巨大複合体の構造的特徴と挙動をモデリングするには理想的である。たとえば、2013年、研究チームはNAMDを使ってHIVカプシド(ウイルスゲノムを取り囲むタンパク質の殻)のモデルを作った。これは、1300を超える同じタンパク質で構成されており、それらがカゴのような構造を作っている。そのような構造は、ホストセルに入るまでウイルスを守るためのものである。そのシミュレーションは、6400万を超える原子の相互作用を説明し、スーパーコンピュータ、Blue Watersの利用が必要だった。新しい研究もBlue Watersを利用した。今度は、コンピュータ顕微鏡の解像度向上のためである。
NAMDソフトウエアは、個々の原子の挙動を表現するために設計されている。しかし特定の化学的相互作用や反応に関わる個々の原子は、常に他所の同等の原子と同じ挙動とは限らない。それらがどのように違うかを理解するには、サブアトミックフォースの働きをよく見る必要がある。これは、分子の動的領域では特に重要である、たとえば、化学結合ができたら壊れたりする領域である。
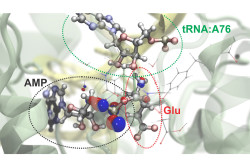
新しいアプローチのデモンストレーションで研究チームは、転移RNA(tRNA)の化学的挙動をシミュレートした。これは、遺伝情報をタンパク質に移す際に重要な役割を果たす分子。NAMDを使い、研究チームは、特殊タンパク質がアミノ酸をtRNAに乗せる瞬間のtRNAの全般的な分子構造をモデル化した。研究チームは、複合体の2つのサイトを、もっと焦点を絞った量子力学的アプローチを必要とする領域に分けた。
2つの領域の相互作用のサブアトミックシミュレーションにより研究チームは、4つの異なるシナリオのシミュレーションを走らせることができた。これにより、tRNAは、細胞内と同じように機能することができる。このシミュレーションによって、4つの可能な化学的経路の一つが、ほかのものよりもエネルギー的に起こりやすい、つまり起こりがちであることがわかった。
(詳細は、www.illinois.edu)


















